
Fig1.
La nature a, au fil du temps, écrit le livre le plus long et complexe à ce jour : le code génétique.
L’Adénine, de Thymine, de Guanine et de Cytosine forment des mots définissants notre existence, qui nous sommes et notre histoire.
Ce livre encore globalement incompris renferme en lui les plus grands secrets, de notre histoire jusqu’à notre plus infime caractéristique.
Le livre est en écriture continuelle, évoluant de réplications cellulaires en mutations génétiques de telle manière que nous sommes tous scribe d’une œuvre éperdument magnifique permettant la vie sous la forme que nous connaissons.
Le scribe que nous sommes ne rédige qu’involontairement ce code, du moins… Ce n’est plus exactement vrai.
CRISPR/Cas, les ciseaux génétiques
Avant tout, il s’agit d’un système de défense immunitaire présent chez 50 à 60% des bactéries et 90% des archées, microorganismes unicellulaires procaryotes, c’est-à-dire des êtres vivants constitués d’une seule cellule unique qui ne comprend ni noyau ni organites, à l’instar des bactéries. (Wikipédia).
Le système est composé de deux parties :
- CRISPR, pour Clustered Regularly Interspaced Short Palindromic Repeats,
- Un groupe de gènes répondants aux mêmes signaux : un opéron Cas.
Ce dernier opéron va être responsable de la synthèse des protéines Cas.
Les virus sont lus, reconnus et découpés pour être rendus inactifs.
Qui dit multitude d’espèces disposant de CRISPR/Cas, dit également multitude de CRISPR/Cas.
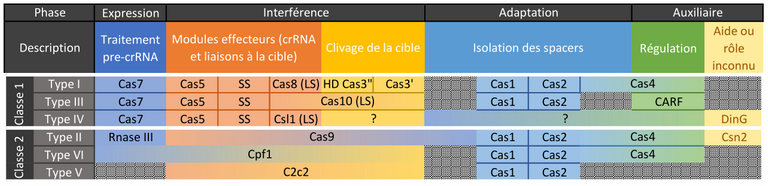
Fig2.
Dans notre cas, le type II est utilisé pour sa « simplicité ».
Lorsqu’une bactérie est infectée par un virus et que cette infection est dite abortive (sans décès de l’organisme), les enzymes de l’adaptation Cas1 et Cas2 (pour le type II) vont inclure le code génétique dans une base de données héréditaire nommée CRISPR-array.
Chaque morceau d’ADN de virus - spacers de 20 nucléotides - est séparé par du code toujours identique qui peut être palindromique (qui se lit dans les deux sens) bien que ce ne soit pas le cas ici pour CRISPR/Cas91,2,3.
On pourrait alors comparer cet array à un télégramme codé comme ceci dans l’ADN :
Virus1. Stop. Virus2. Stop. Virus3. Stop.
Plus tard, si un même phage infecte cette bactérie ou sa descendance, les enzymes de l’interférence (Cas9 ici) vont venir lire l’ADN du phage et s’il correspond à une valeur de l’array, elle le découpe et le rend inactif4,5.
Il faut noter qu’une nouvelle variante de Cas, nommée CasRx permet d’agir sur l’ARN* et non plus sur l’ADN. CasRx ne sera pas abordé ici.*
Détails techniques
Disclaimer : certaines notions abordées le seront succinctement, histoire de ne pas pondre un livre ! :D
Au bout de la séquence du CRISPR-array, se trouve une région appelée Leader Sequence. Elle permet de transcrire cet array en ARN.
Via l’expression de l’ARN, les séquences phagiques vont être exprimées en une longue séquence d’ARN dite pre-crRNA. Celle-ci sera alors reconnue par une protéine Cas (CRISPR-associated protein) synthétisée par l’opéron : une endoribonucléase.
Cette endoribonucléase (RNase) va reconaître chaque répétitions « inter-spacers », cliver l’ARN au niveau de chaque spacers pour produire de petits fragments d’ARN (crRNA) qui iront s’associer à une protéine Cas (endonucléase Cas9).
Ce complexe Cas va ensuite « parcourir » l’ADN du phage jusqu’à ce que le fragment d’ARN se lie à son ADN par complémentarité de base.
L’enzyme produit alors une cassure double brin (cassure des deux brins d’ADN) via le domaine HNH Nuclease Domain (Cyan sur la représentation ci-dessous) et le RuvC Nuclease Domain (en gris).
Dans la nature, il faut un autre ARN en plus du crRNA : le tracrRNA.
Les laboratoires de Jennifer DOUDNA et d’Emmanuelle CHARPENTIER ont travaillé conjointement afin de combiner ces deux ARN en un seul : sgRNA (single guide RNA)1,2. L’opération ne nécessite maintenant plus qu’un seul ARN au lieu de deux.
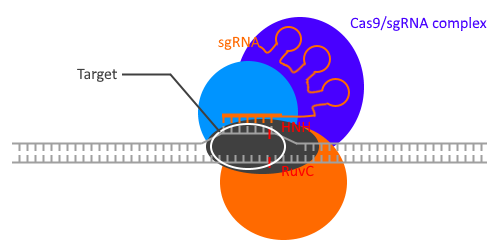
Fig3.
Une fois cette cassure effectuée, l'organisme fait intervenir un mécanisme appelé recombinaison homologue décomposé en 3 étapes :
- Une phase pré-synaptique (le site lésé est reconnu),
- Une phase synaptique (l’organisme cherche un morceau d’ADN similaire (homologue) pour réparer la liaison),
- Une phase post-synaptique (restauration de l’ADN sur le modèle de l’homologue.
Cela implique qu’en injectant un complexe CRISPR/Cas9 avec de l’ADN homologue donné, l’ADN homologue se retrouve génétiquement incorporé dans l’ADN de l’organisme.
Perspectives et évolutions
CRISPR/Cas9 présente de nombreux avantages. Universel, il agit comme un outil ne demandant qu’un ARN là où les anciennes pratiques demandaient une synthèse protéique et enzymatique spécifique longue et coûteuse.
Les techniques évoluent même vers une simplification puisqu’il est possible de sélectionner plusieurs cibles par un seul sgRNA via l’utilisation de séquences universelles10.
Il est aussi possible de modifier Cas9 (en « dead » Cas9) pour inhiber ou favoriser la transcription d’un gène comme le rapportent Vijai Singh, et al., (2016). En modifiant les domaines enzymatiques Ruv-C (D10A) et HNH (H840A), l’enzyme se lie à l’ADN mais ne peut plus couper l’ADN, elle va interférer avec l’expression des gènes**.
C’est ce que l’on nomme CRISPR-Interference (ou CRISPRi) pour une inhibition (via dCas9-KRAB pour les mammifères) et CRISPR-activation (CRISPRa) pour une activation (via par exemple, le système multiplasmide SAM).
Maintenant que l’Homme peut modifier l’ADN, tout est envisageable. Il sera alors possible de guérir les maladies génétiques comme la mucoviscidose, l’hémophilie ou la drépanocytose.
CRISPR/Cas9 est aussi envisagé comme possible traitement contre les cellules tumorales en inversant les tares.
Sans évoquer la possibilité de dommages collatéraux, le complexe s’ouvre à de nombreuses questions éthiques notamment vis-à-vis de l’eugénisme avec la modification d’embryons humains.
Les possibilités sont tellement importantes que le gouvernement américain s’inquiète d’un futur terrorisme d’un nouveau genre, pouvant désactiver certaines fonctions vitales ou causer d’importantes mutations9.
Références
[1.] Conférence « The CRISPR-Cas9 revolution genome engineering: lessons learned from bacteria » par Emmanuelle CHARPENTIER.
[2.] Conférence « Genome Engineering and the Future of Human Health » par Jennifer DOUDNA.
[3.] Amitai G, Sorek R. 2016. CRISPR-Cas adaptation: insights into the mechanism of action. Nat. Rev. Microbiol. 14(2):67–76.
[4.] Barrangou R, Fremaux C, Deveau H, Richards M, Boyaval P, et al. 2007. CRISPR provides acquired resistance against viruses in prokaryotes. Science 315(5819):1709–12.
[5.] Makarova KS, Grishin NV, Shabalina SA, Wolf YI, Koonin EV. 2006. A putative RNA-interferencebased immune system in prokaryotes: computational analysis of the predicted enzymatic machinery, functional analogies with eukaryotic RNAi, and hypothetical mechanisms of action. Biol. Direct 1:7.
[6.] Jörg Renkawitz, Claudio A. Lademann et Stefan Jentsch, « Mechanisms and principles of homology search during recombination », Nature Reviews. Molecular Cell Biology, vol. 15, no 6, 1er juin 2014, p. 369–383.
[7.] Vijai Singh, et al., Exploring the potential of genome editing CRISPR-Cas9 technology, 2016.
[8.] Bikard et al., 2013; Qi et al., 2013
[9.] [https://motherboard.vice.com/en_us/article/yp3xaj/obamas-science-advisors-are-worried-about-future-crispr-terrorism](https://motherboard.vice.com/en_us/article/yp3xaj/obamas-science-advisors-are-worried-about-future-crispr-terrorism)
[10.] Toon Swings, et al., CRISPR-FRT targets shared sites in a knock-out collection for off-the-shelf genome editing, 2018.
Table des illustrations
Fig1. Illustration personnelle, CC BY-ND Clément POIRET. Adobe Photoshop & Illustrator.
Fig2. Illustration personnelle, CC BY-SA Clément POIRET. Microsoft Excel.
Fig3. Illustration personnelle, CC BY-SA Clément POIRET. Paint(dot)Net <3.
Pour aller plus loin

Bannière par @nitesh9 et @rocking-dave
Merci aux communautés SteemSTEM et FrancoSTEM pour leur aide et leur soutient ! <3
Très intéressant!
J'attends avec impatience les résultats des premières expérimentations sur la création de thérapies géniques type CAR-T allogéniques contre les cancers. Les biotechs française Cellectis et belge Celyad sont à la pointe d cette technologie mais pas avant quelques années. Elles sont en stade précliniques je crois.
Upvoté à 100%
Merci :)
C'est sûr, dans les prochaines années ce type de manipulation génétique pourra en aider beaucoup ! Ça renforce mon intention d'aller en doctorat pour participer à la recherche ! 😂
Hi @clement.poiret!
Your post was upvoted by utopian.io in cooperation with steemstem - supporting knowledge, innovation and technological advancement on the Steem Blockchain.
Contribute to Open Source with utopian.io
Learn how to contribute on our website and join the new open source economy.
Want to chat? Join the Utopian Community on Discord https://discord.gg/h52nFrV